DNA 메틸화
"오늘의AI위키"의 AI를 통해 더욱 풍부하고 폭넓은 지식 경험을 누리세요.
1. 개요
DNA 메틸화는 DNA 염기에 메틸기가 부착되는 화학 반응으로, 진핵생물에서 유전자 발현 조절, 세포 분화, 발생, 질병 등 다양한 생물학적 과정에 관여한다. 포유류에서는 주로 CpG 부위의 시토신에 메틸화가 일어나며, 식물에서는 CpG, CpHpG, CpHpH 부위에서 시토신이 메틸화될 수 있다. DNA 메틸화는 DNA 메틸전이효소(DNMT)에 의해 촉매되며, DNMT1은 메틸화 패턴 유지, DNMT3A와 DNMT3B는 새로운 메틸화 패턴 확립에 관여한다. DNA 메틸화는 유전자 발현 조절, 유전체 각인, X 염색체 불활성화, 전이인자 억제 등 다양한 기능을 수행하며, 암, 동맥경화증, 심부전, 노화, 신경 발달 질환 등 여러 질병과 관련이 있다. DNA 메틸화 연구 방법으로는 질량 분석법, 메틸화 특이적 PCR, 비스울파이트 시퀀싱 등이 사용되며, 곤충, 균류, 세균 등 다양한 생물에서도 DNA 메틸화가 발견된다.
더 읽어볼만한 페이지
- 후성유전학 - 메틸화
메틸화는 메틸기를 분자에 첨가하는 과정으로, 생물학에서 유전자 발현 조절 등 생명현상에 관여하고 유기화학에서 유기 합성에 활용되며, 다양한 생물종에 존재하는 중요한 생리적 과정이다. - 후성유전학 - 후성유전체
후성유전체는 DNA 염기 서열 변화 없이 유전자 발현이나 표현형에 영향을 주는 유전적 변화로, DNA 메틸화, 히스톤 변형, 비코딩 RNA를 이용한 유전자 침묵 등의 유형이 있으며, 세포 기능과 발달 조절, 질병 이해 및 치료, 개인 건강 관련 요인 규명에 기여한다. - DNA - 미토콘드리아 DNA
미토콘드리아 DNA(mtDNA)는 세포 호흡에 필수적인 유전 정보를 담고 있는 미토콘드리아 내의 이중 가닥 환상 DNA로서, 동물의 경우 약 16,000개의 염기쌍과 37개의 유전자로 구성되며, 모계 유전, 높은 돌연변이율, 질병 및 노화와의 연관성 등의 특징을 가진다. - DNA - 이중 나선
이중 나선은 DNA의 구조로서, 두 개의 폴리뉴클레오티드 사슬이 반대 방향으로 배열되어 오른손잡이 나선 구조를 이루며, 상보적인 염기쌍이 수소 결합으로 연결되고 주구와 부구라는 고랑을 가지며 다양한 형태가 존재한다.
| DNA 메틸화 | |
|---|---|
| 지도 정보 | |
| 기본 정보 | |
| 유형 | 생물학적 과정 |
| 관련 생물학적 과정 | 유전체 조절 |
| 특징 | |
| 설명 | DNA에서 시토신 또는 아데닌 염기의 화학적 변형으로, 유전체 조절에 중요한 역할. |
| 시토신 메틸화 | 포유류에서 가장 일반적인 형태, 주로 CpG 부위에서 발생. |
| 아데닌 메틸화 | 일부 박테리아와 진핵생물에서 발견, 최근 포유류에서도 보고됨. |
| 효소 | DNA 메틸트랜스퍼라제 (DNMT)에 의해 촉매됨. |
| 효과 | 유전자 발현 조절, 염색체 안정성 유지, 세포 분화 및 발생 조절 등 다양한 생물학적 과정에 영향. |
| 연구 | 후성유전체 연구의 중요한 부분, 암 및 기타 질병 연구에 활용. |
| 시토신 메틸화 상세 정보 | |
| 위치 | 주로 시토신 염기의 5번 탄소 (5mC) CpG 디뉴클레오타이드에서 자주 발생 |
| 기능 | 유전자 발현 억제 염색체 구조 조절 DNA 복제 및 복구 과정에 관여 |
| 패턴 | 세포 유형 특이적 발생 단계에 따라 변화 |
| 연구 방법 | 바이설파이트 시퀀싱 메틸화 특이적 PCR (MSP) 면역침강 |
| 관련 질병 | 암 발달 장애 신경 질환 |
| 아데닌 메틸화 상세 정보 | |
| 위치 | 아데닌 염기의 N6 위치 (6mA) |
| 기능 | DNA 복제 전사 조절 DNA 손상 복구 |
| 연구 | 액체 크로마토그래피-질량 분석법 (LC-MS/MS) |
| 종별 메틸화 특징 | |
| 포유류 | 시토신 메틸화 (5mC)가 주요 형태 CpG 부위에서 주로 발견 배아 발생, 조직 특이적 발현에 중요 |
| 식물 | 시토신 메틸화 (5mC)가 일반적 CpG, CHG, CHH 부위에서 발견 (H는 A, C 또는 T) |
| 곰팡이 | 일부 종에서 시토신 메틸화 발견 예를 들어, Neurospora crassa는 RIP (Repeat-Induced Point Mutation) 과정에서 소량의 메틸화된 DNA를 가짐 Dictyostelium discoideum은 DNA 메틸화가 거의 없음 |
| 효모 | Saccharomyces cerevisiae와 Schizosaccharomyces pombe는 DNA 메틸화가 없음 |
| 초파리 | Drosophila melanogaster에서 시토신 메틸화 발견 |
| 선충 | Caenorhabditis elegans에서 메틸화된 시토신 및 하이드록시메틸시토신이 발견됨 |
| 추가 정보 | |
| 역사적 연구 | Physarum polycephalum에서 초기 DNA 메틸화 연구 진행 |
2. 역사
DNA 메틸화는 1948년 롤린 호치키스(Rollin Hotchkiss)에 의해 처음 발견되었다.[136] 1970년대에는 제한 효소와 DNA 메틸화의 관계가 밝혀졌다. 1980년대에는 DNA 메틸화가 유전자 발현 조절에 중요한 역할을 한다는 사실이 밝혀졌다. 1990년대 이후 DNA 메틸화와 관련된 다양한 연구가 진행되고 있으며, 암을 비롯한 여러 질병과의 연관성이 밝혀지고 있다.[137]
DNA 메틸화는 DNA 염기 중 시토신(C) 또는 아데닌(A)에 메틸기(-CH3)가 붙는 화학 반응이다. 포유류에서는 주로 CpG 부위(시토신과 구아닌이 인산기로 연결된 부위)의 시토신에 메틸화가 일어난다. 식물에서는 CpG, CpHpG, CpHpH 부위의 시토신에 메틸화가 일어난다 (H는 A, C, T 중 하나).[76] DNA 메틸화는 DNA 메틸전이효소(DNMT)에 의해 촉매된다.
척추동물의 DNA 메틸화 지형은 다른 생물에 비해 매우 특이하다. 포유류의 경우, 체세포 CpG 이중 핵산염기의 약 75%가 메틸화되어 있으며, DNA 메틸화는 특정 위치에서 특이적으로 제외되어야 하는 기본 상태로 나타난다.[19] [16][20] 반면, 대부분의 식물, 무척추동물, 균류 또는 원생생물의 게놈은 메틸화된 영역과 메틸화되지 않은 영역이 번갈아 나타나는 "모자이크" 메틸화 패턴을 보이며, 특정 게놈 요소만을 표적으로 한다.[21][22]
3. 생화학적 특징
메틸화된 시토신(C) 잔기는 시간이 지남에 따라 자발적으로 탈아미노화되어 티민(T) 잔기를 형성한다. 따라서 CpG 이중 핵산염기는 꾸준히 TpG 이중 핵산염기로 탈아미노화되며, 이는 인간 게놈에서 CpG 이중 핵산염기의 과소 표현(예상 빈도의 21%만 존재)으로 나타난다.[23] 반면, 메틸화되지 않은 C 잔기의 자발적 탈아미노화는 우라실(U) 잔기를 생성하며, 이는 세포에 의해 신속하게 인식되고 복구된다.
4. 생물학적 기능
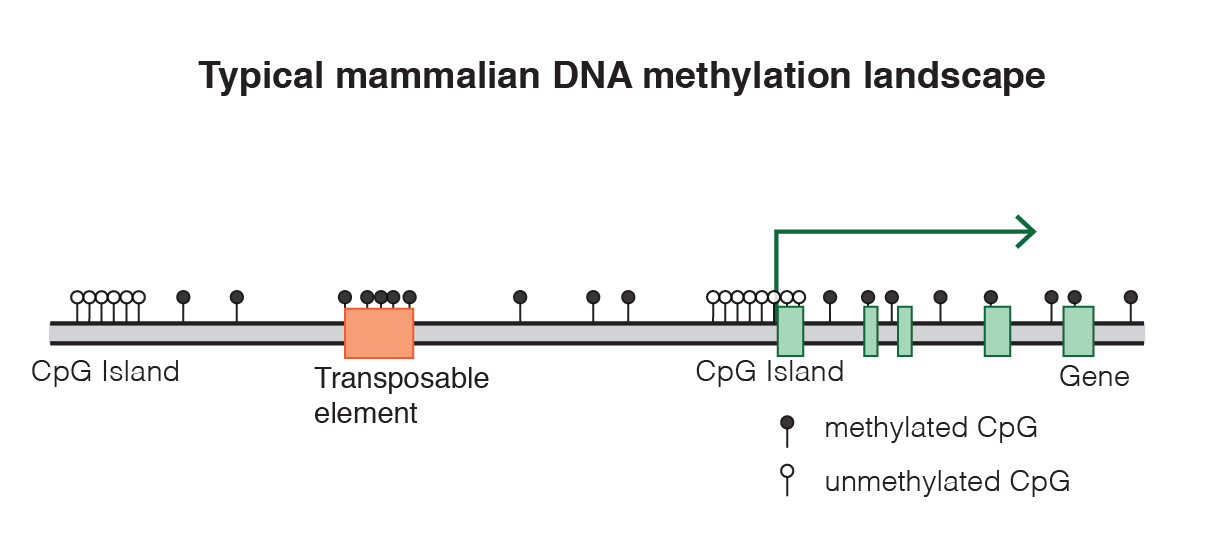
포유류 게놈에서 높은 CpG 메틸화는 자발적 돌연변이 빈도를 증가시키기 때문에 진화적 비용이 따른다. 시토신의 아미노기 손실은 높은 빈도로 발생하며, 메틸화 여부에 따라 결과가 다르다. 메틸화된 C 잔기는 시간이 지남에 따라 자발적으로 탈아미노화되어 T 잔기를 형성한다. 따라서 CpG 이중 핵산염기는 꾸준히 TpG 이중 핵산염기로 탈아미노화되며, 이는 인간 게놈에서 CpG 이중 핵산염기의 과소 표현(예상 빈도의 21%만 존재)으로 입증된다.[23] 반면, 메틸화되지 않은 C 잔기의 자발적 탈아미노화는 U 잔기를 생성하며, 이는 세포에 의해 신속하게 인식되고 복구된다.
DNA 메틸화는 시토신(Cytosine)의 피리미딘(Pyrimidine) 고리 5번 탄소 원자 또는 아데닌(Adenine)의 퓨린(Purine) 고리 6번 질소 원자에 메틸기(Methyl group)가 첨가되는 반응이다(시토신과 아데닌은 DNA를 구성하는 4가지 염기 중 2가지이다). 이러한 변형은 세포 분열을 거쳐도 유전된다. 일반적으로 DNA 메틸화는 접합자 형성 과정에서 제거되고 발생 과정에서의 이후 세포 분열을 통해 재구축된다. 그러나 최근 연구에 따르면 접합자에서는 메틸기의 완전한 제거보다는 메틸기의 수산화(Hydroxylation)가 일어나는 것으로 나타났다.[136]
DNA 메틸화는 또한 거의 모든 종류의 암 발생에 매우 중요한 역할을 한다.[137] DNA 메틸화는 정상적인 발생에 필수적이며, 각인 유전 및 X 염색체 불활성화, 반복 서열의 억제, 발암 등 많은 중요 단계와 관련이 있다.
비메틸화 CpG는 종종 많은 유전자의 5' 조절 영역에 존재하는 '''CpG 섬'''이라고 불리는 클러스터로 모여 있다. 암 등 많은 질병 과정에서 유전자 프로모터인 CpG 섬이 비정상적인 과메틸화를 받아 결과적으로 세포 분열에 의한 자세포에 전달되는 유전자 침묵이 일어난다. DNA 메틸화의 변화는 암 발생의 중요한 요소로 인식되고 있다. 과메틸화가 프로모터와 관련되어 유전자(종양 억제 유전자) 침묵을 일으키는 반면, 저메틸화는 일반적으로 초기 발생하는 염색체 불안정성이나 각인 상실과 관련이 있다. 그러나 저메틸화는 후성유전학적 치료의 표적이 될 수도 있다.[144]
지금까지의 연구에 따르면, 인간의 장기 기억 유지가 DNA 메틸화에 의해 조절된다는 것이 시사되고 있다.[145][146]
4. 1. 유전자 발현 조절
DNA 메틸화는 DNA에 메틸기가 첨가되는 반응으로, 주로 시토신의 5번 탄소 위치에 메틸기가 붙는 경우 유전자 발현이 감소하는 효과가 있다. DNA 메틸화는 초기 진핵생물 조상에서부터 존재했을 가능성이 높으며, 분석된 거의 모든 생물체에서 프로모터 영역의 메틸화는 유전자 발현 억제와 관련이 있다.[21][28] 활발하게 전사되는 유전자의 CpG가 풍부한 프로모터는 메틸화되지 않지만, 전사가 억제된 유전자가 반드시 메틸화된 프로모터를 가지는 것은 아니다.
DNA 메틸화는 두 가지 방식으로 유전자 전사에 영향을 줄 수 있다.[32]
특히, MeCP2의 손실은 레트 증후군과 관련이 있으며, MBD2는 암에서 과메틸화된 유전자의 전사 침묵을 매개한다.
DNA 메틸화는 전이인자의 영구적인 침묵을 보장하는 데 중요한 역할을 한다.[33] 전이인자 조절은 DNA 메틸화의 가장 오래된 기능 중 하나로 여겨지며, DNA 메틸화가 이 목적으로 진화했다는 주장도 있다.[34][35]
유전자 본체 메틸화는 스플라이싱 조절[37] 및 유전자 내 전사 단위 활성 억제[38]에 관여하는 것으로 보인다. 유전자 본체 메틸화는 H3K36 메틸화와 밀접하게 관련되어 있으며, 이는 크로마틴을 응축시켜 암호성 시작 부위의 활성화를 방지한다.[39]
4. 2. 세포 분화 및 발생
DNA 메틸화는 세포 분화 및 발생 과정에서 핵심적인 역할을 수행한다. 발생 초기 단계에서 DNA 메틸화 패턴이 재확립되며, 이는 세포의 운명을 결정하는 데 중요한 역할을 한다.[40] 특정 유전자의 메틸화 패턴은 세포 유형에 따라 다르게 나타나며, 이는 조직 특이적인 유전자 발현을 조절한다.[40]
포유류에서는 세대 간에 DNA 메틸화 패턴이 대부분 지워졌다가 다시 확립된다. 부모로부터 물려받은 거의 모든 메틸화는 생식세포형성 과정과 초기 배아발생 과정에서 탈메틸화와 재메틸화를 통해 지워진다.[40] 초기 배아 발생에서 탈메틸화는 수정란, 상실배, 포배에서 일어나며, 이후 착상 단계에서 메틸화가 일어나 CpG 섬은 메틸화로부터 보호된다. 이는 하우스키핑 유전자가 모든 세포에서 발현되도록 한다.[40] 착상 후 단계에서는 메틸화 패턴이 단계 및 조직 특이적으로 변화하며, 각 세포 유형을 정의하는 변화는 오랜 기간 안정적으로 유지된다.[40] 쥐의 사지 돌기 연구에서는 발생 과정에서 DNA 메틸화의 역동적인 특성이 관찰되었으며, 이는 기관 형성 과정에서 메틸화의 복잡한 조절과 재생 의학에 대한 잠재적 의미를 강조한다.[41]
DNA 메틸화는 유전체 각인과 X 염색체 불활성화에서 단일 대립 유전자 침묵 유지에 중요하며,[42][43] DNA 메틸화 손실은 각인 손실과 체세포에서 Xist의 재발현을 초래한다. 배아 발생 과정에서 생식세포 특이적 발현 유전자를 제외한 소수의 유전자만이 메틸화 상태를 변화시킨다.[44] 분화된 세포에서 DNA 메틸화는 필수적이며, DNA 메틸전이효소 녹아웃은 배아 또는 분만 후 치사율을 유발한다. 반면, 미분화 세포 유형에서는 DNA 메틸화가 불필요하다.
유전체 각인 현상으로 인해 모계 및 부계 게놈은 차별적으로 표시되며, 생식세포를 통과할 때마다 DNA 메틸화 재프로그래밍을 통해 재프로그래밍되어야 한다. 수정 후에는 부계 및 모계 게놈이 탈메틸화 및 재메틸화되지만, 각인된 유전자와 관련된 차별적 메틸화 영역은 제외된다. 이러한 재프로그래밍은 배아의 전능성과 획득된 후생유전학적 변화의 지움에 필요하다.[45]
B 세포 분화 주기 연구에 따르면, 초기 단계부터 분화된 단계까지 저메틸화가 나타나며, 생식 중심 B 세포와 기억 B 세포 단계 사이에서 가장 큰 메틸화 차이가 나타난다. 또한 B 세포 종양과 장수명 B 세포는 DNA 메틸화 서명에서 유사성을 보인다.[18]
DNA 메틸화는 정상적인 발생과 세포 분화에 중요하며, 세포 기억을 통해 유전자 발현 패턴을 안정적으로 변화시키거나 유전자 발현을 감소시킨다.
4. 3. 유전체 각인
유전체 각인은 부모 중 어느 쪽에서 유래한 유전자인지에 따라 유전자 발현이 달라지는 현상이다. 생식세포형성 과정에서 원시 생식세포는 부모에게서 받은 DNA 메틸화 패턴을 지우고, 전달하는 부모의 성별에 따라 다시 설정한다. 수정 후, 부계와 모계 게놈은 다시 탈메틸화와 재메틸화 과정을 거친다. (단, 각인된 유전자와 관련된 차별적으로 메틸화된 영역은 제외). 이러한 재프로그래밍은 새로 형성된 배아의 전능성과 획득된 후생유전학적 변화를 제거하는데 필요하다.[45]
DNA 메틸화는 유전체 각인과 X 염색체 불활성화에서 단일 대립 유전자 침묵을 유지하는 데 중요하다.[42][43] 발현되는 대립 유전자와 침묵되는 대립 유전자는 메틸화 상태가 다르며, DNA 메틸화가 소실되면 각인이 소실되고 체세포에서 Xist의 재발현이 나타난다.
4. 4. X 염색체 불활성화
포유류 암컷은 X 염색체를 두 개 가지고 있는데, 이 중 하나가 불활성화되는 현상을 X 염색체 불활성화라고 한다. 이는 DNA 메틸화와 밀접하게 관련되어 있으며, 유전자 발현의 균형을 유지하는 데 중요한 역할을 한다.
DNA 메틸화는 정상적인 발생에 필수적이며, X 염색체 불활성화와 같은 여러 중요한 단계와 관련이 있다.[136]
4. 5. 전이인자 억제
DNA 메틸화는 전이인자의 영구적인 억제를 보장하는 데 매우 적합하다.[33] 전이인자 조절은 동물, 식물 및 여러 원생생물이 공유하는 DNA 메틸화의 가장 오래된 기능 중 하나이다.[34] DNA 메틸화가 바로 이 목적, 즉 전이인자 억제를 위해 진화했다는 주장도 있다.[35]
4. 6. 게놈 확장
DNA 메틸화는 시간이 지남에 따라 숙주의 게놈에 통합된 바이러스나 기타 유해 요소의 유전자 발현을 억제한다.[137] 특히 전이인자의 DNA 메틸화는 게놈 확장과 관련이 있는 것으로 알려져 있다.
5. DNA 메틸화와 질병
DNA 메틸화는 정상적인 발생 과정에 필수적이며, 각인 유전, X 염색체 불활성화, 반복 서열 억제, 발암 등 여러 중요한 단계와 관련이 있다. 포유류에서는 CpG 부위의 60~90%가 메틸화되어 있으며,[141][142] 메틸화된 시토신은 자발적인 탈아미노화 반응으로 티민으로 변이될 수 있다. 비메틸화 CpG는 주로 유전자의 5' 조절 서열에 위치한 CpG 섬에 모여 있다.
DNA 메틸화는 암을 비롯한 여러 질병과 연관되어 있다. 암과 같은 많은 질병 과정에서 유전자 프로모터 CpG 섬의 비정상적인 과메틸화는 세포 분열을 통해 딸세포에 전달되는 유전자 사일렌싱을 유발한다.[46] DNA 메틸화의 변화는 암 발생의 중요한 요소로, 과메틸화는 프로모터와 관련되어 종양 억제 유전자 침묵을 일으키는 반면, 저메틸화는 일반적으로 초기 발생하는 염색체 불안정성이나 각인 상실과 관련이 있다.[47]
DNA 메틸화는 두 가지 방식으로 유전자 전사에 영향을 미친다. 첫째, DNA 메틸화 자체가 전사 인자의 결합을 물리적으로 방해할 수 있다. 둘째, 메틸화된 DNA는 메틸-CpG 결합 도메인 단백질(MBD)과 결합하고, MBD 단백질은 히스톤 탈아세틸화 효소 및 기타 크로마틴 재구성 단백질을 유전자좌에 모집하여 불활성화된 크로마틴을 형성한다.[144] 특히, MeCP2의 결손은 레트 증후군과 관련이 있으며, MBD2는 암에서 과메틸화 유전자의 전사 억제를 매개한다.
인간의 장기 기억 유지가 DNA 메틸화에 의해 조절된다는 연구 결과도 있다.[145][146]
5. 1. 암
암세포에서는 정상 세포와 다른 DNA 메틸화 패턴이 나타난다. 일반적으로 종양 억제 유전자의 과메틸화와 암유전자의 저메틸화가 관찰된다.[49] 종양 억제 유전자 프로모터의 CpG 섬 과메틸화는 해당 유전자의 발현을 억제하여 암 발생을 촉진할 수 있다.[46] 반면, 암유전자의 저메틸화는 유전자 발현을 증가시켜 암 발생을 촉진할 수 있다.[47]암으로 진행되면서 수백 개의 유전자가 침묵되거나 활성화되는데, 이 중 상당 부분은 DNA 메틸화 변화 때문이다.[46] 특히, DNA 복구 유전자의 프로모터 CpG 섬 메틸화는 암 진행에 중요한 역할을 하는 것으로 보인다.[46] 마이크로RNA 발현 변화 또한 CpG 섬의 과/저메틸화를 통해 암 발생에 영향을 미친다.
DNA 메틸화는 전사 인자가 DNA에 결합하는 것을 방해하거나, 메틸화 CpG 결합 도메인 단백질(MBD)을 통해 히스톤 탈아세틸화 효소 등을 유도하여 불활성화된 크로마틴을 형성함으로써 유전자 전사를 조절한다.
DNA 메틸화 패턴은 특정 암의 진단 및 예후 예측에 활용될 수 있다. 예를 들어, 유방암 및 자궁암에서 에스트로겐 수용체 유전자의 메틸화는 해당 유전자의 발현 감소와 관련이 있다.[148]
한국에서는 DNA 메틸화를 기반으로 한 암 조기 진단 및 치료 기술 개발 연구가 활발히 진행되고 있다. 특히, 혈액 샘플에서 특정 유전자의 메틸화 패턴을 분석하여 암을 조기에 진단하는 기술 개발에 대한 연구가 활발하다.
5. 2. 동맥경화증
DNA 메틸화와 같은 후성유전적 변형은 동맥경화증을 포함한 심혈관 질환과 관련이 있다. 동물 동맥경화증 모델에서 혈관 조직뿐만 아니라 단핵구, 혈구와 같은 혈구는 유전자 특이적 과메틸화 영역을 포함한 전반적인 저메틸화를 나타낸다. DNA 메틸화 다형성은 병변이 관찰되기 전에 존재하기 때문에 동맥경화증의 조기 바이오마커로 사용될 수 있으며, 이는 조기 검출 및 위험 예방을 위한 도구를 제공할 수 있다.[50]DNA 메틸화 다형성의 표적이 되는 두 가지 세포 유형은 단구와 림프구이며, 이들은 전반적인 저메틸화를 경험한다. 이러한 전반적인 저메틸화의 한 가지 제안된 메커니즘은 상승된 호모시스테인 수치로 인한 고호모시스테인혈증으로, 이는 심혈관 질환의 알려진 위험 요소이다. 높은 혈장 호모시스테인 수치는 DNA 메틸전이효소를 억제하여 저메틸화를 유발한다. DNA의 저메틸화는 평활근 세포 증식을 변화시키고, 내피 세포 기능 장애를 유발하며, 염증 매개체를 증가시키는 유전자에 영향을 미치는데, 이는 모두 동맥경화성 병변 형성에 중요하다.[51] 높은 수치의 호모시스테인은 또한 에스트로겐 수용체 알파(ERα) 유전자의 프로모터 영역에서 CpG 섬의 과메틸화를 초래하여 발현을 감소시킨다.[52] ERα는 성장 억제제로 작용하여 평활근 세포가 휴지 상태를 유지하도록 하기 때문에 동맥경화증으로부터 보호한다.[53] 따라서 ERα 프로모터의 과메틸화는 내막 평활근 세포가 과도하게 증식하여 동맥경화성 병변의 발생에 기여할 수 있게 한다.[54]
동맥경화증에서 메틸화 상태 변화를 경험하는 또 다른 유전자는 일탄소 운반체(MCT3)이며, 이는 혈관 평활근 세포를 포함한 많은 세포 유형에서 젖산염 및 기타 케톤체의 수송을 담당하는 단백질을 생성한다. 동맥경화증 환자의 경우 엑손 2의 CpG 섬 메틸화가 증가하여 MCT3 단백질 발현이 감소한다. MCT3의 발현 감소는 젖산염 수송을 손상시키고 평활근 세포 증식을 크게 증가시켜 동맥경화성 병변에 더 기여한다. 탈메틸화제인 데시타빈(5-aza-2-deoxycytidine)을 사용한 생체 외 실험에서 엑손 2 CpG 섬의 모든 과메틸화 부위가 처리 후 탈메틸화됨에 따라 용량 의존적 방식으로 MCT3 발현을 유도하는 것으로 나타났다. 이는 아직 인간에 대한 연구는 수행되지 않았지만 동맥경화증 치료를 위한 새로운 치료제가 될 수 있다.[55]
5. 3. 심부전
심부전 환자의 심장에서는 특정한 후성유전학적 변화가 확인되었다. 이는 질병의 원인에 따라 다를 수 있다. 예를 들어, 허혈성 심부전에서는 DNA 메틸화 변화가 유전자 발현 변화와 관련이 있으며, 이는 알려진 심장 대사 변화와 관련된 유전자 발현을 조절할 수 있다.[56] 다른 형태의 심부전(예: 당뇨병성 심근병증)과 동반 질환(예: 비만)에 대해서도 이러한 메커니즘이 얼마나 흔한지 조사해야 한다. 가장 두드러지게, 심부전 환자의 심장에서 이러한 DNA 메틸화 변화는 인종과 사회경제적 지위와 관련이 있으며, 이는 유전자 발현 변화에 추가적인 영향을 미치고,[57] 개인의 심부전 치료 방식에도 영향을 줄 수 있다.5. 4. 노화
인간과 다른 포유류에서 DNA 메틸화 수준은 조직과 세포 유형의 나이를 정확하게 추정하는 데 사용될 수 있으며, 정확한 후성적 시계를 형성한다.[58]종단 연구에서 쌍둥이 어린이의 경우 5세에서 10세 사이에 유전적 영향보다는 환경적 영향으로 인해 메틸화 패턴의 차이가 나타났다.[59] 노화 과정 동안 DNA 메틸화는 전반적으로 감소한다.[49]
신생아, 26세, 103세 개인의 CD4+ T 세포의 완전한 DNA 메틸롬을 분석한 연구에서 메틸화의 손실이 나이에 비례한다는 사실이 관찰되었다.[60] 신생아와 비교하여 백세인의 DNA에서 관찰된 저메틸화 CpG는 모든 게놈 구획(프로모터, 유전자간, 인트론 및 엑손 영역)을 포함했다.[60] 그러나 에스트로겐 수용체, p16, 인슐린 유사 성장 인자 2,[49] ELOVL2[61] 및 FHL2[62]를 포함한 일부 유전자는 나이가 들면서 고메틸화된다.
5. 5. 신경 발달 질환
레트 증후군은 메틸-CpG 결합 단백질 2(MeCP2) 유전자의 돌연변이로 인해 발생하는 신경 발달 질환이다.[144] MeCP2는 메틸화된 DNA에 결합하여 유전자 발현을 조절하는 단백질로, 레트 증후군 환자에서는 이러한 기능에 이상이 생긴다.5. 6. 기타 질환
DNA 메틸화는 암 외에도 다양한 질병 과정과 관련이 있다. 많은 질병에서 유전자 프로모터 CpG 섬은 비정상적인 과메틸화를 획득하여 전사 침묵을 유발하는데, 이는 세포 분열 후 딸세포에 상속될 수 있다.[46] DNA 메틸화의 변화는 암 발생의 중요한 구성 요소로, 일반적으로 저메틸화는 염색체 불안정성 및 각인 상실과 관련이 있고, 과메틸화는 프로모터와 관련되어 유전자(종양 억제 유전자) 침묵을 유발할 수 있지만, 후성유전 요법의 표적이 될 수 있다.[47]6. DNA 메틸전이효소 (DNMT)
포유류에서 DNA 메틸화를 촉매하는 효소는 DNA 메틸전이효소(DNMT)이다. 포유류에는 DNMT1, DNMT3A, DNMT3B, DNMT3L 등 여러 종류의 DNMT가 존재한다.[72]
DNMT1은 DNA 복제 과정에서 기존의 메틸화 패턴을 딸 가닥에 복사하여 유지하는 역할을 하는 유지 메틸전이효소로 제안되었다. DNMT1이 결핍된 쥐 모델은 배아 9일째에 치사하는데, 이는 포유류 세포 발달에 DNMT1 활성이 필수적임을 보여준다.
DNMT3A와 DNMT3B는 발생 초기에 새로운 메틸화 패턴을 확립하는 *de novo* 메틸전이효소로 생각된다. DNMT3L은 촉매 활성이 없는 단백질이지만, *de novo* 메틸전이효소와 결합하여 DNA 결합 능력을 높이고 활성을 돕는다.[72]
쥐와 생쥐는 Muroidea 설치류의 공통 조상에서 탠덤 중복(tandem duplication)에 의해 ''Dnmt3b''의 파라로그(paralog)로 진화한 DNMT3C라는 세 번째 기능적 *de novo* 메틸전이효소 효소를 가지고 있다. DNMT3C는 초기 정자 형성 중에 전좌 가능 요소(transposable element)의 프로모터 메틸화를 촉매하며, 이는 이들의 후생유전적 억제와 수컷 생식력에 필수적인 것으로 나타났다.[73][74]
DNMT2 (TRDMT1)는 DNA가 아닌 아스파르트산 tRNA의 안티코돈 루프에 존재하는 시토신-38을 메틸화한다.[75]
| 효소 | 기능 |
|---|---|
| DNMT1 | DNA 복제 과정에서 기존 메틸화 패턴을 유지 |
| DNMT3A | 발생 과정에서 새로운 메틸화 패턴 확립 |
| DNMT3B | 발생 과정에서 새로운 메틸화 패턴 확립 |
| DNMT3L | 촉매 활성은 없지만, DNMT3A와 DNMT3B의 활성을 도움 |
| DNMT2 (TRDMT1) | 아스파르트산 tRNA의 안티코돈 루프에 존재하는 시토신-38을 메틸화, DNA 메틸화와 관련 없음 |
7. DNA 메틸화 연구 방법
DNA 메틸화는 현재 과학 연구에서 사용되는 다음과 같은 분석법으로 검출할 수 있다.[101]
전통적인 방법
- 메틸화 특이적 PCR (MSP): DNA와 아황산나트륨의 화학 반응을 기반으로, CpG 이중 핵산염기의 비메틸화된 시토신을 우라실(UpG)로 전환한다. 메틸화된 시토신은 전환되지 않으므로, 관심 CpG 부위에 상보적인 프라이머를 설계하여 PCR 후 메틸화 여부를 판단한다.[102]
- 제한 랜드마크 게놈 스캐닝: 제한 효소가 메틸화 및 비메틸화 CpG 부위를 구별하여 인식하는 점을 이용하는 분석법이다. 현재는 거의 사용되지 않으며, HELP 분석법과 개념적으로 유사하다.
- 메틸 민감성 서던 블롯팅: HELP 분석법과 유사하게 제한 효소 절단을 사용하지만, 서던 블롯팅 기술을 이용하여 유전자 특이적 메틸화 차이를 조사한다. 프로브 결합 부위 근처의 국소 메틸화를 평가한다.
- 고해상도 융해 분석(HRM 또는 HRMA): PCR 후 분석 기술이다. 아황산나트륨으로 비메틸화 시토신을 우라실로 전환하고, 메틸화된 시토신은 보존한다. 메틸화 및 비메틸화 템플릿 모두 증폭되도록 프라이머를 설계하여 PCR 증폭 후, 융해 온도 차이를 통해 정량적 메틸화 검출을 수행한다.[106][107]
- 메틸화 민감성 단일 뉴클레오티드 프라이머 신장 분석법(msSNuPE): 검출할 뉴클레오티드 바로 앞(5')에 어닐링되는 내부 프라이머를 사용한다.[110]
- 파이로시퀀싱: 비스울파이트 처리된 DNA를 파이로시퀀싱한다. 일반적인 포워드 프라이머로 앰플리콘을 만들고, 비오틴화된 리버스 프라이머로 선택 유전자를 PCR한다. 파이로시퀀서가 지정된 순서대로 뉴클레오티드를 추가하며 DNA를 변성, 분석한다. 불일치 발생 시 기록되며, 불일치 DNA 비율을 통해 CpG 섬당 메틸화 비율을 계산한다.
차세대 염기서열 분석 (NGS) 기반 방법
- 전장 게놈 비스울파이트 시퀀싱(BS-Seq): 게놈 DNA를 아황산나트륨으로 전환 후, 차세대 시퀀싱 플랫폼에서 시퀀싱한다. 얻어진 시퀀스를 참조 게놈에 정렬, 우라실 전환으로 인한 불일치를 통해 CpG 이중 핵산염기 메틸화 상태를 결정한다.
- 축소 표현 비스울파이트 시퀀싱(RRBS): 메틸롬의 약 10%를 대상으로 하며, 참조 게놈이 필요하다. 이후 샘플 다중화를 높이고 게놈의 더 작은 부분을 시퀀싱하는 프로토콜들이 개발되었다. EpiGBS는 Illumina 시퀀싱 레인 하나에서 96개 샘플 다중화가 가능하며, 참조 게놈이 필요 없다. Watson과 Crick 판독값으로 새로운 참조를 구축하여 SNP, SMP 집단 스크리닝을 동시에 수행한다.
- 효소적 메틸-시퀀싱(EM-시퀀싱): 비스울파이트 시퀀싱과 유사하나, APOBEC 및 TET2 효소를 사용하여 비메틸화 시토신을 우라실로 탈아미노화한다. 비스울파이트 처리보다 DNA 손상이 적다.[103]
- 일루미나 메틸화 분석법: 어레이 하이브리드화를 통해 특정 위치(locus)의 DNA 메틸화를 측정한다. 비스울파이트 처리 DNA를 "BeadChip" 프로브에 하이브리드화하고, 표지된 프로브로 단일 염기 신장을 통해 메틸화 상태를 결정한다.[111] 2016년, 85만 개 이상 메틸화 부위를 조사하는 Infinium MethylationEPIC BeadChip이 출시되었다.[112]
기타 방법
- 질량 분석법: DNA 메틸화 검출에 매우 민감하고 신뢰할 수 있지만, 염기서열 맥락 정보를 제공하지 않아 기능 연구에는 한계가 있다.
- HELP 분석법: 제한 효소가 메틸화 및 비메틸화 CpG DNA 부위를 구별하여 인식하고 절단하는 능력을 이용한다.
- GLAD-PCR 분석법: 메틸화된 DNA만 가수분해하는 새로운 유형의 효소인 부위 특이적 메틸화 지향 DNA 엔도뉴클레아제를 사용한다.
- 칩상 ChIP 분석법: MeCP2 등 DNA 메틸화 관련 단백질에 결합하는 항체를 사용한다.
- 메틸화 DNA 면역 침전법(MeDIP): 크로마틴 면역 침전법과 유사하게, 면역 침전법으로 메틸화 DNA 단편을 분리하여 DNA 마이크로어레이(MeDIP-칩) 또는 DNA 시퀀싱(MeDIP-seq)에 사용한다.
- DNA 아데닌 메틸전이효소 활성에 대한 분자 브레이크 라이트 분석법: 형광체와 켄처로 표지된 올리고뉴클레오티드에서 완전 메틸화(아데닌 메틸화)된 GATC 부위에 대한 제한 효소 DpnI의 특이성을 이용한다. 아데닌 메틸전이효소가 올리고뉴클레오티드를 메틸화하면 DpnI 기질이 되고, DpnI 절단 시 형광이 증가한다.[104][105]
- 메틸CpG 결합 단백질(MBP) 및 메틸 결합 도메인(MBD) 포함 융합 단백질을 사용하여 천연 DNA를 메틸화 및 비메틸화 분획으로 분리한다. 각 분획의 표적 양을 정량화하여 CpG 섬 메틸화 비율을 결정한다. 스크립션 기반 검출은 FFPE 조직에서 매우 민감한 검출을 가능하게 한다.
- 고대 DNA 메틸화 재구성: 고대 DNA 샘플에서 고해상도 DNA 메틸화를 재구성한다. 메틸화 시토신은 티민으로, 비메틸화 시토신은 우라실로 분해되는 자연적 과정을 이용한다. 분해 신호 비대칭성으로 네안데르탈인, 데니소바인 전체 메틸화 지도를 재구성했다.[108] 2019년, DNA 메틸화 데이터에서 형태 특징을 추론하는 새로운 방법이 발표되었다. 하향 조절 유전자를 단일 유전자 질환 표현형과 연결하여 DNA 메틸화 지도로부터 해부학적 특징을 약 85% 정확도로 재구성했다.[109]
8. DNA 메틸화와 관련된 기타 내용
DNA 메틸화는 시토신의 피리미딘 고리 5번 탄소 원자 또는 아데닌의 퓨린 고리 6번 질소 원자에 메틸기(Methyl group)가 첨가되는 반응이다. 이러한 변형은 세포 분열을 거쳐도 유전된다. 일반적으로 DNA 메틸화는 접합자 형성 과정에서 제거되고 발생 과정에서의 이후 세포 분열을 통해 재구축되지만, 최근 연구에 따르면 접합자에서는 메틸기의 완전한 제거보다는 메틸기의 수산화(Hydroxylation)가 일어나는 것으로 나타났다.[136]
DNA 메틸화는 세포가 "자신이 어디에 있는지"를 기억(세포 기억)할 수 있도록 안정적으로 유전자 발현 패턴을 변화시키거나 유전자 발현을 감소시킨다. 예를 들어, 이자의 랑게르한스섬이 되도록 프로그램된 세포는 지속적으로 신호를 받지 않더라도 랑게르한스섬으로 남는다. 또한 DNA 메틸화는 시간이 지남에 따라 숙주의 게놈(Genome)에 통합된 바이러스(Virus)나 기타 유해 요소의 유전자 발현을 억제하며, 크로마틴(Chromatin) 구조의 기반을 형성하여 단일 DNA 서열에서 다세포 생물에 필요한 수많은 특징을 형성할 수 있게 한다. DNA 메틸화는 거의 모든 종류의 암(Cancer) 발생에 매우 중요한 역할을 한다.[137]
DNA 메틸화는 DNA에 메틸기가 첨가되는 것을 수반하며, 시토신의 피리미딘 고리 5번 탄소 원자에 메틸기가 붙으면 유전자 발현 감소라는 특이적인 효과가 있다. 시토신 5번 위치의 메틸화는 조사된 모든 척추동물(Vertebrate)에서 발견된다. 성체 체세포 조직에서 DNA 메틸화는 일반적으로 CpG 부위(시토신-포스포디에스터 결합-구아닌)에서 발생하며, 비CpG 메틸화는 배아줄기세포(Embryonic stem cell)에서 널리 퍼져 있다.[138][139][140]
포유류에서 모든 CpG 부위의 60~90%는 메틸화되어 있다.[141][142] 메틸화된 시토신 잔기는 자발적으로 아미노기가 제거되어 티민 잔기가 되는데, CpG 이중 핵산염기는 TpG 이중 핵산염기로 변이된다. 이는 인간 게놈에서 CpG 이중 핵산염기의 출현 빈도가 낮다는 점에서 명확하다(CpG 이중 핵산염기는 예상 빈도의 21%만 존재한다).[143] 반면, 비메틸화 시토신의 자발적인 탈아미노화에서는 우라실 잔기가 생성되지만, 이 변이는 세포에 의해 빠르게 인식되어 복구된다.
비메틸화 CpG는 종종 많은 유전자의 5' 조절 영역에 존재하는 '''CpG 섬'''이라고 불리는 클러스터로 모여 있다. 암 등 많은 질병 과정에서 유전자 프로모터인 CpG 섬이 비정상적인 과메틸화를 받아 결과적으로 세포 분열에 의한 자세포에 전달되는 유전자 사일렌싱이 일어난다. DNA 메틸화의 변화는 암 발생의 중요한 요소로 인식되고 있다. 과메틸화가 프로모터와 관련되어 종양 억제 유전자의 사일렌싱을 일으키는 반면, 저메틸화는 일반적으로 초기 발생하는 염색체 불안정성이나 각인 상실과 관련이 있다. 그러나 저메틸화는 후성유전학적 치료의 표적이 될 수도 있다.[144]
DNA 메틸화는 두 가지 방법으로 유전자 전사에 영향을 미친다. 하나는 DNA의 메틸화 자체가 물리적으로 전사 단백질의 유전자 결합을 방해하는 것이고, 다른 하나는 메틸화 DNA가 메틸화 CpG 결합 도메인 단백질(MBD)과 결합하는 것이다. MBD 단백질은 히스톤을 변형하는 히스톤 탈아세틸화 효소 및 기타 크로마틴 재구성 단백질 등 추가적인 단백질을 모집하여 불활성화된 크로마틴(사일런트 크로마틴)을 형성한다. 특히, MeCP2(MeCP2)의 결손은 레트 증후군과 관련이 있으며, MBD2(MBD2)는 암에서 과메틸화 유전자의 전사 사일렌싱을 매개한다.
지금까지의 연구에 따르면, 인간의 장기 기억 유지가 DNA 메틸화에 의해 조절된다는 것이 시사되고 있다.[145][146]
8. 1. 곤충의 DNA 메틸화
다양한 곤충목은 DNA 메틸화 패턴이 다양하게 나타나는데, 초파리에서는 거의 검출되지 않는 수준에서부터 나비목에서는 낮은 수준, 그리고 노린재목과 일부 바퀴벌레(Blattella asahinai)에서는 모든 CG 부위의 최대 14%까지)에서는 더 높은 수준으로 나타난다.[82]꿀벌에서 기능적 DNA 메틸화가 발견되었다.[83][84] DNA 메틸화 표지는 주로 유전자 본체에 있으며, DNA 메틸화 기능에 대한 현재의 의견은 대체 스플라이싱을 통한 유전자 조절이다.[85]
초파리(Drosophila melanogaster)의 DNA 메틸화 수준은 거의 검출되지 않는다.[86] 초파리 DNA에 적용된 민감한 방법은 전체 시토신의 0.1~0.3% 범위의 수준을 시사한다.[87] 2014년 연구에 따르면 초파리에서 나타나는 낮은 수준의 메틸화는 "특정 짧은 모티프에서 나타나며 DNMT2 활성과는 무관하다"는 것을 발견했다.[88] 게다가, 고감도 질량 분석법[89]을 통해 초파리 배아 발생 초기 단계에서 낮은(0.07%) 수준이지만 유의미한 아데닌 메틸화 수준이 존재함을 입증했다.
8. 2. 균류의 DNA 메틸화
많은 균류는 시토신 메틸화 수준이 낮지만(0.1~0.5%), 다른 균류는 게놈의 최대 5%까지 메틸화될 수 있다.[90] 이 값은 종 간과 같은 종의 균주 간에 모두 변하는 것으로 보인다.[91] 또한 DNA 메틸화가 균류에서 상태 특이적 유전자 발현 조절에 관여할 수 있다는 증거도 있다. 그러나 초고감도 질량 분석법을 사용하여 250 아토몰의 검출 한계에서 ''빵효모(Saccharomyces cerevisiae)'' 또는 ''분열효모(Schizosaccharomyces pombe)''와 같은 단세포 효모 종에서는 DNA 메틸화가 확인되지 않았으며, 이는 효모가 이러한 DNA 변형을 가지고 있지 않음을 나타낸다.[9]맥주 효모(''사카로마이세스속(Saccharomyces)''), 분열 효모(''쉬조사카로마이세스속(Schizosaccharomyces)''), 및 ''아스페르길루스 플라부스(Aspergillus flavus)''[92]는 검출 가능한 DNA 메틸화가 없지만, 모형 사상균인 ''뉴로스포라 크라사(Neurospora crassa)''는 잘 특징지어진 메틸화 시스템을 가지고 있다.[93] 여러 유전자가 ''뉴로스포라''의 메틸화를 조절하며, DNA 메틸전이효소 ''dim-2''의 돌연변이는 모든 DNA 메틸화를 제거하지만 성장이나 유성 생식에는 영향을 미치지 않는다. ''뉴로스포라'' 게놈에는 반복 DNA가 거의 없지만, 메틸화의 절반은 전이인자 잔류물과 동원체 DNA를 포함한 반복 DNA에서 발생한다. DNA 메틸화효소 결핍 유전적 배경에서 다른 중요한 현상을 평가할 수 있는 능력은 ''뉴로스포라''를 DNA 메틸화 연구에 중요한 시스템으로 만든다.
''디크티오스텔륨 디스코이데움''(Dictyostelium discoideum)에서는 DNA 메틸화가 거의 나타나지 않으며,[94] 시토신의 약 0.006%에서만 발생하는 것으로 보인다.[6] 반대로, ''피사룸 폴리세팔룸''(Physarum polycephalum)에서는 DNA 메틸화가 널리 분포하며,[95] 5-메틸시토신이 전체 시토신의 최대 8%를 차지한다.[4]
8. 3. 세균 및 고세균의 DNA 메틸화
세균의 제한-변형 시스템은 아데닌 또는 시토신 메틸화를 매개하는데, 이 시스템에서 특정 DNA 서열은 게놈 전체에 걸쳐 주기적으로 메틸화된다.[97] 메틸라제는 특정 서열을 인식하고 그 서열 내 또는 근처의 염기 중 하나를 메틸화하는 효소이다. 세포에 도입된 외래 DNA(이런 방식으로 메틸화되지 않음)는 서열 특이적 제한 효소에 의해 분해되고 절단된다. 세균 게놈 DNA는 이러한 제한 효소에 의해 인식되지 않는다. 원래 DNA의 메틸화는 일종의 원시적인 면역 체계 역할을 하여 세균이 박테리오파지 감염으로부터 자신을 보호할 수 있도록 한다.''대장균'' DNA 아데닌 메틸트랜스퍼라제(Dam)는 제한/변형 시스템에 속하지 않는 약 32 kDa의 효소이다. ''대장균'' Dam의 표적 인식 서열은 GATC이며, 메틸화는 이 서열(G meATC)의 아데닌 N6 위치에서 발생한다. 이 부위의 양쪽에 있는 세 개의 염기쌍도 DNA-Dam 결합에 영향을 미친다. Dam은 오류 수정, DNA 복제 시기 및 유전자 발현을 포함한 여러 가지 주요 세균 과정에서 여러 역할을 한다. DNA 복제의 결과로, ''대장균'' 게놈의 GATC 부위 상태는 완전히 메틸화된 상태에서 반메틸화된 상태로 변한다. 이는 새로운 DNA 가닥에 도입된 아데닌이 메틸화되지 않았기 때문이다. 재메틸화는 2~4초 이내에 발생하며, 이 기간 동안 새로운 가닥의 복제 오류가 복구된다. 메틸화 또는 메틸화의 부재는 세포의 복구 기구가 주형 가닥과 새로 합성된 가닥을 구별할 수 있게 해주는 표지이다. 세균에서 Dam 활성을 변경하면 자발적 돌연변이율이 증가하는 것으로 나타났다. 특정 다른 DNA 복구 효소가 없는 dam 돌연변이체에서는 세균 생존력이 손상되어 DNA 복구에서 Dam의 역할에 대한 추가적인 증거를 제공한다.
더 오랫동안 반메틸화 상태를 유지하는 DNA 영역 중 하나는 복제 기점이며, 이곳에는 GATC 부위가 풍부하다. 이것은 DNA 복제 시기를 조절하는 세균 메커니즘의 중심이다. SeqA는 복제 기점에 결합하여 이를 격리하고 따라서 메틸화를 방지한다. 반메틸화된 복제 기점은 비활성이기 때문에 이 메커니즘은 세포 주기당 DNA 복제를 한 번으로 제한한다.
특정 유전자(예: ''대장균''의 필리 발현을 코딩하는 유전자)의 발현은 유전자 오페론의 프로모터 영역에 있는 GATC 부위의 메틸화에 의해 조절된다. DNA 복제 직후 세포의 환경 조건은 Dam이 프로모터 영역에 근접하거나 멀리 떨어진 영역의 메틸화를 차단할지 여부를 결정한다. 메틸화 패턴이 생성되면 필리 유전자 전사는 DNA가 다시 복제될 때까지 켜기 또는 끄기 위치로 고정된다. ''대장균''에서 이러한 필리 오페론은 요로 감염에서 병독성에 중요한 역할을 한다.
반면에, DNA 시토신 메틸라제는 CCAGG 및 CCTGG 부위를 표적으로 하여 C5 위치(C meC(A/T)GG)에서 시토신을 메틸화한다. 다른 메틸라제 효소인 EcoKI는 AAC(N6)GTGC 및 GCAC(N6)GTT 서열에서 아데닌의 메틸화를 유발한다.
''클로스트리디오이데스 디피실레''에서는 표적 모티프 CAAAAA에서의 DNA 메틸화가 질병 전파의 핵심 단계인 포자 형성뿐만 아니라 세포 길이, 생물막 형성 및 숙주 감염에도 영향을 미치는 것으로 나타났다.[98]
9. 결론 및 전망
DNA 메틸화는 유전자 발현 조절, 세포 분화, 발생, 질병 등 다양한 생명 현상에 관여하는 중요한 후성유전학적 기전이다. DNA 메틸화 연구는 암을 비롯한 여러 질병의 진단 및 치료 기술 개발에 기여할 수 있을 것으로 기대된다.
한국의 전망: 한국에서는 DNA 메틸화 연구가 활발히 진행되고 있으며, 특히 암 조기 진단 및 치료 기술 개발 분야에서 괄목할 만한 성과가 기대된다.
참조
[1]
논문
The occurrence of 6-methylaminopurine in deoxyribonucleic acids
//www.ncbi.nlm.nih.g[...]
1958-04-01
[2]
논문
Rare bases in animal DNA
1970-01-01
[3]
논문
DNA methylation in thermophilic bacteria: N6-methylcytosine, 5-methylcytosine, and N6-methyladenine.
//www.ncbi.nlm.nih.g[...]
1985-01-01
[4]
논문
Methylation of the deoxyribonucleic acid of Physarum polycephalum at various periods during the mitotic cycle
1970-12-01
[5]
논문
Lack of 5-methylcytosine in Dictyostelium discoideum DNA
1991-07-01
[6]
논문
The ''Dictyostelium discoideum'' genome lacks significant DNA methylation and uncovers palindromic sequences as a source of false positives in bisulfite sequencing
2023-04-01
[7]
논문
Trace analysis of methylated and hydroxymethylated cytosines in DNA by isotope-dilution LC-MS/MS: first evidence of DNA methylation in Caenorhabditis elegans
2015-01-01
[8]
논문
Molecular biology. Methylation talk between histones and DNA
2001-12-01
[9]
논문
Cytosine DNA methylation is found in Drosophila melanogaster but absent in Saccharomyces cerevisiae, Schizosaccharomyces pombe, and other yeast species
2014-04-01
[10]
논문
N6-methyladenine: the other methylated base of DNA
2006-03-01
[11]
논문
DNA methylation on N(6)-adenine in mammalian embryonic stem cells
2016-04-01
[12]
웹사이트
Uracil in DNA: error or signal?
http://www.sciencein[...]
[13]
논문
Reviving the RNA World: An Insight into the Appearance of RNA Methyltransferases
2016-01-01
[14]
논문
De novo methylation of MMLV provirus in embryonic stem cells: CpG versus non-CpG methylation
2002-05-01
[15]
논문
Allele-specific non-CpG methylation of the Nf1 gene during early mouse development
2001-12-01
[16]
논문
Human DNA methylomes at base resolution show widespread epigenomic differences
2009-11-01
[17]
논문
Global epigenomic reconfiguration during mammalian brain development
2013-08-01
[18]
논문
Whole-genome fingerprint of the DNA methylome during human B cell differentiation
2015-07-01
[19]
논문
DNA methylation: an introduction to the biology and the disease-associated changes of a promising biomarker
2010-01-01
[20]
논문
DNA-binding factors shape the mouse methylome at distal regulatory regions
2011-12-01
[21]
논문
Genome-wide evolutionary analysis of eukaryotic DNA methylation
2010-05-01
[22]
논문
CpG methylation is targeted to transcription units in an invertebrate genome
2007-05-01
[23]
논문
Initial sequencing and analysis of the human genome
2001-02-01
[24]
논문
CpG-rich islands and the function of DNA methylation
1986-05-15
[25]
논문
CpG islands in vertebrate genomes
1987-07-01
[26]
논문
Orphan CpG islands identify numerous conserved promoters in the mammalian genome
2010-09-01
[27]
논문
A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters
2006-01-01
[28]
논문
Conservation and divergence of methylation patterning in plants and animals
2010-05-01
[29]
논문
Lineage-specific polycomb targets and de novo DNA methylation define restriction and potential of neuronal progenitors
2008-06-01
[30]
논문
Distribution, silencing potential and evolutionary impact of promoter DNA methylation in the human genome
2007-04-01
[31]
논문
Function and information content of DNA methylation
2015-01-01
[32]
논문
Genome-wide conserved consensus transcription factor binding motifs are hyper-methylated
2010-09-01
[33]
논문
Genome-wide analysis in the mouse embryo reveals the importance of DNA methylation for transcription integrity
2020-06-01
[34]
논문
Dnmt1-independent CG methylation contributes to nucleosome positioning in diverse eukaryotes
2014-03-01
[35]
논문
Cytosine methylation and the ecology of intragenomic parasites
1997-08-01
[36]
논문
DNA methylation enables transposable element-driven genome expansion
2020-08-00
[37]
논문
The alternative role of DNA methylation in splicing regulation
2015-05-00
[38]
논문
Conserved role of intragenic DNA methylation in regulating alternative promoters
2010-07-00
[39]
논문
Histone H3 methylation by Set2 directs deacetylation of coding regions by Rpd3S to suppress spurious intragenic transcription
2005-11-00
[40]
논문
Programming of DNA methylation patterns
https://zenodo.org/r[...]
2012-07-00
[41]
논문
Global DNA methylation and chondrogenesis of rat limb buds in a three-dimensional organ culture system
2022-07-00
[42]
논문
Loss of methylation activates Xist in somatic but not in embryonic cells
1995-10-00
[43]
논문
Role for DNA methylation in genomic imprinting
1993-11-00
[44]
논문
Targets and dynamics of promoter DNA methylation during early mouse development
2010-12-00
[45]
논문
Reprogramming DNA methylation in the mammalian life cycle: building and breaking epigenetic barriers
2013-01-00
[46]
논문
Metabolic recoding of epigenetics in cancer
2018-05-00
[47]
논문
Specific gene hypomethylation and cancer: new insights into coding region feature trends
2009-04-00
[48]
서적
Epigenetics: A Reference Manual
Caister Academic Press
[49]
논문
Epigenetic alterations in aging
2010-08-00
[50]
논문
DNA methylation polymorphisms precede any histological sign of atherosclerosis in mice lacking apolipoprotein E
2004-07-00
[51]
논문
Increased homocysteine and S-adenosylhomocysteine concentrations and DNA hypomethylation in vascular disease
2003-08-00
[52]
논문
Hypermethylation of estrogen receptor-alpha gene in atheromatosis patients and its correlation with homocysteine
2009-10-00
[53]
논문
DNA methylation and atherosclerosis
2002-08-00
[54]
논문
Methylation of the estrogen receptor-alpha gene promoter is selectively increased in proliferating human aortic smooth muscle cells
2000-04-00
[55]
논문
Inactivation of monocarboxylate transporter MCT3 by DNA methylation in atherosclerosis
2005-08-00
[56]
논문
Genome-wide DNA methylation encodes cardiac transcriptional reprogramming in human ischemic heart failure
2019-03-00
[57]
논문
Racial and socioeconomic disparity associates with differences in cardiac DNA methylation among men with end-stage heart failure
2021-05-00
[58]
논문
DNA Methylation Clocks in Aging: Categories, Causes, and Consequences
2018-09-00
[59]
논문
A longitudinal study of epigenetic variation in twins
2010-08-00
[60]
논문
Distinct DNA methylomes of newborns and centenarians
2012-06-00
[61]
웹사이트
The lipid elongation enzyme ELOVL2 is a molecular regulator of aging in the retina
https://onlinelibrar[...]
2020-02-00
[62]
웹사이트
Impact of age, BMI and HbA1c levels on the genome-wide DNA methylation and mRNA expression patterns in human adipose tissue and identification of epigenetic biomarkers in blood
https://academic.oup[...]
2015-04-10
[63]
논문
Acute exercise remodels promoter methylation in human skeletal muscle
2012-03-00
[64]
논문
A six months exercise intervention influences the genome-wide DNA methylation pattern in human adipose tissue
2013-06-00
[65]
논문
Physical activity and global genomic DNA methylation in a cancer-free population
2011-03-00
[66]
논문
Dynamic DNA methylation controls glutamate receptor trafficking and synaptic scaling
2016-05-00
[67]
논문
Epigenetic regulation and chromatin remodeling in learning and memory
2017-01-00
[68]
논문
Memory consolidation for contextual and auditory fear conditioning is dependent on protein synthesis, PKA, and MAP kinase
1999-00-00
[69]
논문
DNA methylation changes in plasticity genes accompany the formation and maintenance of memory
2016-01-00
[70]
논문
Experience-dependent epigenomic reorganization in the hippocampus
2017-07-00
[71]
논문
DNA Methylation and Establishing Memory
2022
[72]
웹사이트
Review on DNA Methylation
http://www.methods.i[...]
[73]
논문
The DNA methyltransferase DNMT3C protects male germ cells from transposon activity
2016-11-00
[74]
논문
rahu is a mutant allele of Dnmt3c, encoding a DNA methyltransferase homolog required for meiosis and transposon repression in the mouse male germline
2017-08-00
[75]
논문
Methylation of tRNAAsp by the DNA methyltransferase homolog Dnmt2
2006-01-00
[76]
논문
Dynamics and function of DNA methylation in plants
2018-08-00
[77]
논문
DNA methylation in Arabidopsis has a genetic basis and shows evidence of local adaptation
2015-05-00
[78]
논문
Locus-specific control of asymmetric and CpNpG methylation by the DRM and CMT3 methyltransferase genes
2002-12-00
[79]
논문
Widespread natural variation of DNA methylation within angiosperms
2016-09-00
[80]
논문
Genome-wide evolutionary analysis of eukaryotic DNA methylation
2010-05-00
[81]
논문
RNA-directed DNA methylation in Arabidopsis
2002-12-00
[82]
논문
Evolution of DNA Methylation across Insects
2017-03-00
[83]
논문
Functional CpG methylation system in a social insect
2006-10-00
[84]
서적
Physiological and Molecular Mechanisms of Nutrition in Honey Bees
[85]
논문
RNA interference knockdown of DNA methyl-transferase 3 affects gene alternative splicing in the honey bee
2013-07-00
[86]
논문
The two-dimensional restriction analysis of Drosophila DNAs: males and females
1981-05-00
[87]
논문
DNA methylation in Drosophila melanogaster
2000-11-00
[88]
논문
Genome methylation in D. melanogaster is found at specific short motifs and is independent of DNMT2 activity
2014-05-00
[89]
논문
N6-methyladenine DNA modification in Drosophila
2015-05-00
[90]
논문
DNA methylation in the fungi
1984-07-00
[91]
논문
A comparison of DNA methylation levels in selected isolates of higher fungi
[92]
논문
Bisulfite sequencing reveals that Aspergillus flavus holds a hollow in DNA methylation
2012-00-00
[93]
논문
The methylated component of the Neurospora crassa genome
2003-04-00
[94]
논문
Lack of 5-methylcytosine in Dictyostelium discoideum DNA
1991-07-00
[95]
논문
Methjylation in Physarum DNA
1980-07-00
[96]
논문
The Epigenomic Landscape of Prokaryotes
2016-02-00
[97]
논문
Bacterial Epigenomics: Coming of Age
2021-08-00
[98]
논문
Epigenomic characterization of Clostridioides difficile finds a conserved DNA methyltransferase that mediates sporulation and pathogenesis
2020-01-00
[99]
논문
The dam and dcm strains of Escherichia coli--a review
1994-05-00
[100]
웹사이트
Making unmethylated (dam-/dcm-) DNA
http://www.neb.com/n[...]
[101]
논문
Crime investigation through DNA methylation analysis: methods and applications in forensics.
2018-01-00
[102]
논문
Optimizing methodologies for PCR-based DNA methylation analysis
2013-10-00
[103]
논문
Efficient and accurate determination of genome-wide DNA methylation patterns in Arabidopsis thaliana with enzymatic methyl sequencing
2020-10-07
[104]
논문
Kinetic analysis of Yersinia pestis DNA adenine methyltransferase activity using a hemimethylated molecular break light oligonucleotide
2007-08-00
[105]
논문
Hairpin fluorescence DNA probe for real-time monitoring of DNA methylation
2007-02-00
[106]
논문
Methylation-sensitive high resolution melting (MS-HRM): a new approach for sensitive and high-throughput assessment of methylation
[107]
논문
Quantitative evaluation of DNA methylation by optimization of a differential-high resolution melt analysis protocol
2009-07-00
[108]
논문
Reconstructing the DNA methylation maps of the Neandertal and the Denisovan
2014-05-00
[109]
논문
Reconstructing Denisovan Anatomy Using DNA Methylation Maps
2019-09-00
[110]
논문
Methylation Markers for the Identification of Body Fluids and Tissues from Forensic Trace Evidence
2016-02-01
[111]
웹사이트
Infinium Methylation Assay Interrogate single CpG sites
https://www.illumina[...]
2020-01-10
[112]
웹사이트
Infinium MethylationEPIC Kit Methylation profiling array for EWAS
https://www.illumina[...]
2020-01-10
[113]
논문
An integrated resource for genome-wide identification and analysis of human tissue-specific differentially methylated regions (tDMRs)
2008-09-00
[114]
논문
Potential forensic application of DNA methylation profiling to body fluid identification
2012-01-00
[115]
논문
The human colon cancer methylome shows similar hypo- and hypermethylation at conserved tissue-specific CpG island shores
2009-02-00
[116]
논문
Epigenetic reprogramming in mammalian development
2001-08-00
[117]
논문
Genome-scale DNA methylation maps of pluripotent and differentiated cells
2008-08-00
[118]
논문
Differential methylation of tissue- and cancer-specific CpG island shores distinguishes human induced pluripotent stem cells, embryonic stem cells and fibroblasts
2009-12-00
[119]
논문
Intra-individual change over time in DNA methylation with familial clustering
2008-06-00
[120]
논문
Inter-individual variation of DNA methylation and its implications for large-scale epigenome mapping
2008-06-00
[121]
웹사이트
QDMR: a quantitative method for identification of differentially methylated regions by entropy
http://bioinfo.hrbmu[...]
2013-03-09
[122]
논문
QDMR: a quantitative method for identification of differentially methylated regions by entropy
2011-05-00
[123]
논문
Gene-set analysis is severely biased when applied to genome-wide methylation data
2013-08-00
[124]
논문
Systematic identification and annotation of human methylation marks based on bisulfite sequencing methylomes reveals distinct roles of cell type-specific hypomethylation in the regulation of cell identity genes
2016-01-00
[125]
웹사이트
SMART 2: A Comprehensive Analysis Tool for Bisulfite Sequencing Data
http://fame.edbc.org[...]
2016-00-00
[126]
논문
Molecular approaches for forensic cell type identification: On mRNA, miRNA, DNA methylation and microbial markers
2015-09-00
[127]
논문
DNA methylation and application in forensic sciences
2015-04-00
[128]
논문
Developmental validation studies of epigenetic DNA methylation markers for the detection of blood, semen and saliva samples
2016-07-00
[129]
논문
Principles of DNA methylation and their implications for biology and medicine
2018-09-00
[130]
논문
Sensitive and specific multi-cancer detection and localization using methylation signatures in cell-free DNA
2020-06-00
[131]
논문
Comprehensive human cell-type methylation atlas reveals origins of circulating cell-free DNA in health and disease
2018-11-00
[132]
논문
Prediction of methylated CpGs in DNA sequences using a support vector machine
https://eprints.ucm.[...]
2005-08-00
[133]
논문
CpG island methylation in human lymphocytes is highly correlated with DNA sequence, repeats, and predicted DNA structure
2006-03-00
[134]
논문
Enhancement on the predictive power of the prediction model for human genomic DNA methylation
[135]
논문
CpGIMethPred: computational model for predicting methylation status of CpG islands in human genome
[136]
논문
Reprogramming of the paternal genome upon fertilization involves genome-wide oxidation of 5-methylcytosine
[137]
논문
Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals
[138]
논문
De novo methylation of MMLV provirus in embryonic stem cells: CpG versus non-CpG methylation
[139]
논문
Allele-Specific Non-CpG Methylation of the Nf1 Gene during Early Mouse Development
[140]
논문
Human DNA methylomes at base resolution show widespread epigenomic differences
[141]
논문
Amount and distribution of 5-methylcytosine in human DNA from different types of tissues or cells
[142]
논문
Methylated cytosine and the brain: a new base for neuroscience
[143]
논문
Initial sequencing and analysis of the human genome
[144]
논문
Specific gene hypomethylation and cancer: New insights into coding region feature trends
[145]
논문
Covalent modification of DNA regulates memory formation
2007-03-15
[146]
웹사이트
Memories may be stored on your DNA
http://www.newscient[...]
New Scientist
2008-12-02
[147]
서적
Epigenetics: A Reference Manual
Caister Academic Press
[148]
서적
がんの分子生物学
メディカル・サイエンス・インターナショナル
[149]
논문
ゲノムDNAのメチル化修飾の形成と維持の機構
http://lifesciencedb[...]
2012-12-05
[150]
논문
Methylation of tRNAAsp by the DNA methyltransferase homolog Dnmt2
[151]
논문
Locus-specific control of asymmetric and CpNpG methylation by the DRM and CMT3 methyltransferase genes
[152]
웹사이트
ChromDB::Chromatin Database
http://chromdb.org
2011-04-05
[153]
논문
RNA-directed DNA methylation in Arabidopsis
[154]
논문
DNA methylation in the fungi
[155]
논문
A comparison of DNA methylation levels in selected isolates of higher fungi
Mycological Society of America
[156]
논문
Stage-specific DNA methylation in a fungal plant pathogen
http://jb.asm.org/cg[...]
[157]
논문
The methylated component of the Neurospora crassa genome
[158]
논문
Diverse DNA modification in marine prokaryotic and viral communities
https://doi.org/10.1[...]
2022-01-21
[159]
논문
The DNA methylation landscape of giant viruses
https://www.nature.c[...]
2020-05-27
[160]
논문
The dam and dcm strains of ''Escherichia coli''—a review
[161]
웹사이트
Making unmethylated (dam-/dcm-) DNA
http://www.neb.com/n[...]
2011-04-05
[162]
논문
Shotgun bisulphite sequencing of the Arabidopsis genome reveals DNA methylation patterning.
[163]
논문
Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications.
[164]
논문
BSMAP: whole genome bisulfite sequence MAPping program.
[165]
논문
Kinetic analysis of Yersinia pestis DNA adenine methyltransferase activity using a hemimethylated molecular break light oligonucleotide
[166]
논문
Hairpin fluorescence DNA probe for real-time monitoring of DNA methylation
관련 사건 타임라인
( 최근 20개의 뉴스만 표기 됩니다. )
[친절한 식품 이야기]우리 몸의 시계를 되돌리는 열쇠는 ‘잘 고른’ 한 끼 식사
한반도로 온 벼농사…아열대 벗어난 비밀은? [강석기의 과학풍경]
본 사이트는 AI가 위키백과와 뉴스 기사,정부 간행물,학술 논문등을 바탕으로 정보를 가공하여 제공하는 백과사전형 서비스입니다.
모든 문서는 AI에 의해 자동 생성되며, CC BY-SA 4.0 라이선스에 따라 이용할 수 있습니다.
하지만, 위키백과나 뉴스 기사 자체에 오류, 부정확한 정보, 또는 가짜 뉴스가 포함될 수 있으며, AI는 이러한 내용을 완벽하게 걸러내지 못할 수 있습니다.
따라서 제공되는 정보에 일부 오류나 편향이 있을 수 있으므로, 중요한 정보는 반드시 다른 출처를 통해 교차 검증하시기 바랍니다.
문의하기 : help@durumis.com